Catatan tentang Replikasi, Perbaikan, dan Rekombinasi DNA!
Replikasi DNA:
Replikasi DNA semi-konservatif:
Replikasi DNA adalah fungsi autokatalitik DNA. Ini biasanya terjadi selama fase-S siklus sel ketika kromosom berada dalam bentuk yang sangat panjang. Seperti yang dikemukakan oleh Watson dan Crick, replikasi DNA bersifat semi-konservatif.
Dalam replikasi semi-konservatif, dua utas akan terpisah satu sama lain, mempertahankan integritasnya dan masing-masing akan mensintesis, dari kumpulan nukleotida, utas komplementernya. Hasilnya adalah, molekul yang baru disintesis akan membawa atau melestarikan salah satu dari dua untai dari molekul induk dan untai lainnya akan dirakit baru. Ada cukup bukti untuk membuktikan bahwa DNA beruntai ganda benar-benar bereplikasi dengan metode semi-konservatif.
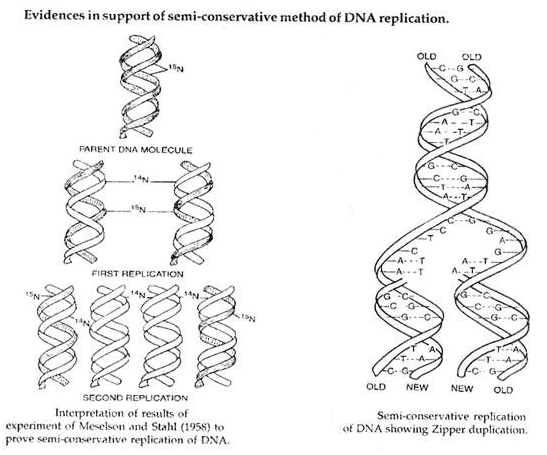
Eksperimen Meselson dan Stahl (1958):
Para pekerja ini membiakkan Escherichia coli dalam media yang mengandung isotop 15 N. Setelah ini direplikasi selama beberapa generasi dalam media itu, kedua untaian DNA ini mengandung 15 N sebagai penyusun purin dan pirimidin. Ketika bakteri dengan 15 N ini dipindahkan ke media kultur yang mengandung 14 N, ditemukan bahwa DNA yang dipisahkan dari bakteri generasi baru memiliki satu untai lebih berat dari yang lain.
Untai yang lebih berat mewakili untai induk dan yang lebih ringan adalah yang baru disintesis dari media kultur, dengan demikian menunjukkan metode replikasi DNA semi-konservatif dan tidak termasuk model sintesis dan replikasi DNA konservatif dan dispersif.
Replikasi konservatif tidak akan menghasilkan molekul DNA dengan konstitusi “hibrid”. Jika replikasi bersifat dispersif, akan ada pergeseran DNA dari “berat ke” ringan “melalui setiap generasi.
Studi selanjutnya telah memverifikasi kesimpulan Meselson dan Stahl bahwa replikasi DNA bersifat semikonservatif dan telah memperluasnya ke banyak organisme lain termasuk tumbuhan dan hewan tingkat tinggi.
Eksperimen autoradiografi Cairn:
Dia menggunakan timin radioaktif atau timidin tritiated. Dengan menumbuhkan E.coli dalam media biakan yang mengandung tritiated thymidine, radioaktivitas tergabung dalam molekul DNA anak.
Dalam autoradiograf, duplikasi molekul DNA menunjukkan garpu replikasi, titik di mana dua rantai menjadi empat. Setelah replikasi pertama, radioaktivitas terlihat tergabung hanya dalam salah satu untai DNA dan kedua untai ditemukan diberi label setelah replikasi kedua. Ini mendukung mode replikasi DNA semi-konservatif.
JH Taylor pada ujung akar Viciafaba (1957) juga mengkonfirmasi metode replikasi DNA semi-konservatif.
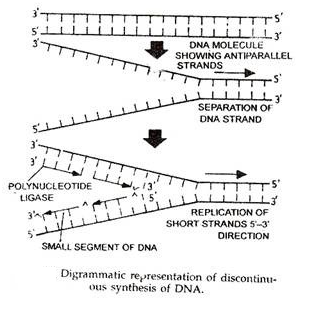
saya. Replikasi DNA terputus-putus:
Okazaki menyarankan bahwa sintesis DNA berlangsung secara bersamaan pada kedua untai DNA dengan menggunakan enzim polimerase DNA yang sama dalam bentuk fragmen kecil yang diisolasi. Segmen ini dikenal sebagai potongan Okazaki dan terdiri dari 1000-2000 nukleotida. Ini digabungkan bersama oleh enzim polinukleotida ligase, melengkapi pembentukan rantai polinukleotida.
Sintesis DNA terputus-putus didukung oleh eksperimen auto-radiografi.
- Replikasi Searah dan Dua Arah DNA:
- Cairns dari eksperimennya menyimpulkan bahwa sintesis DNA dimulai pada titik tetap pada kromosom dan berlangsung dalam satu arah, tetapi eksperimen terbaru menyarankan replikasi dua arah .
Levinthal dan Cairns mengusulkan bahwa selama replikasi, dua helai tidak terpisah sepenuhnya sebelum replikasi. Sebaliknya mereka mulai membuka ritsleting di salah satu ujungnya dan secara bersamaan segmen yang terbuka ritsleting mulai menarik pasangan nukleotida mereka. Dengan cara ini unzip untai DNA asli dan sintesis untai DNA segar berjalan berdampingan.

DNA polimerase:
DNA polimerase adalah enzim utama replikasi DNA. Aktivitasnya pertama kali ditunjukkan oleh Kornberg pada tahun 1956. Ini mengkatalisasi penambahan kovalen deoksiribonukleotida ke 3′-OH dari nukleotida yang sudah ada sebelumnya yang disebut primer.
Enzim DNA polimerase-1 sekarang dianggap sebagai enzim perbaikan DNA daripada enzim replikasi. Enzim ini diketahui memiliki lima situs aktif, yaitu situs templat, situs primer, situs pembelahan atau eksonuklease 5’—>3′, situs nukleosida trifosfat dan situs pembelahan 3’—>5′ (atau 3’†”> 5′ situs eksonuklease).
DNA polimerase I:
Ini terutama terlibat dalam menghilangkan primer RNA dari Okazaki atau fragmen prekursor dan mengisi celah yang dihasilkan karena kapasitas polimerisasi 5’—>3′. Enzim DNA polimerase I juga dapat menghilangkan dimer timin yang dihasilkan akibat penyinaran UV dan mengisi celah akibat eksisi. Ini disebut fungsi proof reading atau editing dari enzim ini.
DNA polimerase-II:
Enzim ini menyerupai DNA polimerase-I dalam aktivitasnya, tetapi merupakan enzim perbaikan DNA dan menghasilkan pertumbuhan dalam arah 5’—> 3′, menggunakan gugus 3′-OH bebas.
DNA polimerase-III:
Ini memainkan peran penting dalam replikasi DNA. Ini adalah enzim multimerik atau holoenzim yang memiliki sepuluh subunit seperti α, β, ε, θ, г, y, δ, δ’, x dan Ψ. Kesepuluh subunit ini diperlukan untuk replikasi DNA in vitro; Namun, semuanya memiliki fungsi yang berbeda. Sebagai contoh, subunit α memiliki 3’—>5′ exonuclease proofreading atau aktivitas editing. Enzim inti terdiri dari tiga subunit – α, β dan θ. Tujuh subunit yang tersisa meningkatkan prosesivitas (prosesivitas berarti kecepatan dan efisiensi yang dengannya DNA polimerase memperluas rantai pertumbuhan).
DNA polimerase eukariotik:
Eukai yotes (misalnya, ragi, hati tikus, sel tumor manusia) ditemukan mengandung lima jenis DNA polimerase berikut:
(i) DNA polimerase α:
Enzim dengan berat molekul relatif tinggi ini juga disebut polimerase sitoplasma atau polimerase besar. Itu ditemukan baik dalam nukleus dan sitoplasma.
(ii) DNA polimerase β:
Enzim ini juga disebut polimerase nuklir atau polimerase kecil dan hanya ditemukan pada vertebrata.
(iii) DNA polimerase y:
Enzim ini disebut polimerase mitokondria dan dikodekan dalam nukleus.
(iv) DNA polimerase δ:
Enzim ini ditemukan dalam sel mamalia dan bergantung pada PCNA untuk proses sintesis DNA (PCNA = proliferating cell nuclear antigen).
(v) DNA polimerase μ:
Itu sebelumnya dikenal sebagai DNA polimerase 5II. Enzim ini tidak bergantung pada PCNA dan terdapat pada sel HeLa mamalia dan ragi pemula.
DNA polimerase a yang besar adalah enzim DNA polimerase yang dominan pada sel eukariotik dan diyakini sejak lama sebagai satu-satunya enzim yang terlibat dalam replikasi DNA. Namun kini satu lagi polimerase, yakni DNA polimerase 8 juga ditemukan terlibat dalam replikasi DNA eukariotik.
Primer DNA:
Enzim ini mengkatalisasi sintesis primer RNA yang merupakan prasyarat untuk inisiasi replikasi DNA pada sebagian besar organisme. Sebelum replikasi DNA sebenarnya dimulai, segmen oligonukleotida RNA pendek, yang disebut primer RNA atau hanya primer, harus disintesis oleh enzim primase DNA menggunakan trifosfat ribonukleosida.
Primer RNA ini disintesis dengan menyalin urutan basa tertentu dari satu untai DNA dan berbeda dari molekul RNA tipikal karena setelah sintesis, primer tetap berikatan hidrogen dengan templat DNA.
Primer memiliki panjang sekitar 10 nukleotida pada eukariota dan dibuat pada interval pada untai tertinggal di mana mereka diperpanjang oleh enzim DNA polimerase untuk memulai setiap fragmen okazaki. Primer RNA ini kemudian dipotong dan diisi dengan DNA dengan bantuan sistem perbaikan DNA pada eukariota.
Pada bakteri, dua enzim berbeda diketahui mensintesis oligonukleotida RNA primer – RNA polimerase (pada untai terdepan) dan DNA primase (pada untai tertinggal).
Polinukleotida Ligase:
Enzim ini merupakan enzim penting baik dalam replikasi DNA maupun dalam perbaikan DNA. DNA ligase mengkatalisis pembentukan hubungan fosfodiester antara gugus 5′-fosforil dari satu nukleotida dan gugus 3-OH dari tetangga terdekat di sisi nick dalam untai DNA; sehingga menyegel torehan dalam untai DNA.
Endonuklease:
Endonuklease, khususnya endonuklease restriksi, juga penting selama replikasi DNA serta perbaikan DNA. Selama replikasi DNA, endounclease dapat menghasilkan torehan pada asal untuk memulai replikasi atau mungkin menginduksi torehan untuk menghasilkan putaran untuk memfasilitasi pelepasan DNA.
Enzim yang Terlibat dalam Pembukaan DNA Helix:
helikase DNA:
Helikase DNA adalah enzim pelepas yang bergantung pada ATP yang mempromosikan pemisahan dua untai induk dan membentuk garpu replikasi yang secara progresif akan menjauh dari asal. Pembukaan heliks DNA cetakan pada garpu replikasi pada prinsipnya dapat dikatalisis oleh dua helikase DNA, bekerja bersama-sama, satu berjalan di sepanjang untai terdepan dan yang lainnya di sepanjang untai tertinggal.
Helix-destabilizing strand (juga disebut protein pengikat DNA untai tunggal atau SSBP):
Di belakang garpu replikasi, untaian DNA tunggal dicegah untuk berputar kembali satu sama lain (atau membentuk loop jepit rambut beruntai ganda di setiap untai tunggal) oleh aksi protein SSB. Protein SSB berikatan dengan untaian DNA yang terbuka tanpa menutupi basa, yang karenanya tetap tersedia untuk proses templating.
Topoisomerase (Girase DNA):
Tindakan helikase memasukkan superkoil positif ke dalam DNA dupleks di depan garpu replikasi. Enzim, yang disebut topoisomerase, mengendurkan superkoil dengan menempel pada dupleks superkoil sementara, memotong salah satu untaian dan memutarnya melalui untaian yang tidak terputus. Nick kemudian disegel kembali.
Salah satu jenis topoisomerase (yaitu, topoisomerase I) menyebabkan pemutusan untai tunggal atau nick yang memungkinkan dua bagian heliks DNA di kedua sisi nick berputar bebas relatif satu sama lain, menggunakan ikatan fosfodiester pada untai yang berlawanan dengan nick sebagai sebuah titik putar. Tipe kedua topoisomerase (yaitu, topoisomerase II) membentuk ikatan kovalen pada kedua heliks DNA heliks pada saat yang sama, membuat untai ganda putus sementara pada heliks.
Replika:
Replika adalah unit DNA di mana tindakan replikasi individu terjadi, yaitu, ia mampu melakukan replikasi DNA terlepas dari segmen DNA lainnya. Oleh karena itu, setiap replika memiliki asal di mana replikasi dimulai dan mungkin memiliki terminal di mana replikasi berhenti.
Kromosom bakteri dan virus biasanya mengandung satu replika/kromosom. Meskipun fag T, memiliki dua asal, satu primer dan satu sekunder, tetapi dengan adanya asal primer, asal sekunder biasanya tidak berfungsi. Pada E. coli titik asal diidentifikasi sebagai lokus oriC genetik.
Asal prokariotik yang lengkap mendukung tiga fungsi berikut: (1) inisiasi replikasi, (2) kontrol frekuensi kejadian inisiasi, dan (3) pemisahan kromosom yang direplikasi ke dalam sel anak.
Asal telah diidentifikasi pada bakteri, ragi, kloroplas dan mitokondria; fitur umum signifikan mereka adalah bahwa mereka kaya A: T yang mungkin penting untuk memudahkan pelepasan selama replikasi. Asal bakteri mengandung banyak situs pendek (<10 bp) berbeda yang diperlukan untuk fungsinya; situs-situs ini terkadang dipisahkan oleh jarak tertentu tetapi tidak oleh urutan tertentu. Situs-situs yang berlokasi khusus ini diperlukan untuk pengikatan berbagai protein yang terlibat dalam replikasi DNA.
Beberapa replika prokariotik memiliki situs spesifik, yang disebut terminus, yang menghentikan gerakan garpu replikasi dan, dengan demikian, mengakhiri replikasi DNA. Kromosom E. coli memiliki dua terminal, disebut T 1 dan T 2 yang terletak sekitar 100 Kb di kedua sisi titik pertemuan garpu replikasi. Setiap terminal khusus untuk satu arah gerakan garpu.
T 1 dan T 2 diatur sedemikian rupa sehingga setiap garpu harus melewati yang lain untuk mencapai ujung yang spesifik. Pengakhiran replikasi memerlukan produk gen tus, yang mungkin mengkode protein yang mengenali T, dan T 2 .
Pada eukariota, setiap kromosom memiliki beberapa replika (misalnya ragi, 500; Drosophila, 3.500; tikus 25.000; Viciafaba, 35.000). Pada titik tertentu selama fase S, hanya sebagian dari replika ini yang mengalami replikasi; setiap replika tampaknya diaktifkan pada waktu tertentu dalam urutan tertentu. Panjang replika eukariotik dapat bervariasi dari 40 Kb pada ragi dan Drosophila hingga sekitar 300 Kb pada Vicia.
Jumlah replika yang terdeteksi tampaknya bervariasi dengan tahap perkembangan dan jenis sel atau jaringan. Hal ini dijelaskan berdasarkan asal spesifik jaringan sehingga beberapa asal aktif di beberapa jaringan, sementara yang lain aktif di beberapa jaringan lain.
Hal ini dicontohkan oleh Drosophila di mana sel-sel embrionik awal memiliki replikasi 10 kali lebih banyak daripada sel-sel somatik dewasa. Bukti yang ada menunjukkan bahwa replika eukariotik tidak memiliki terminii.
Kesetiaan Replikasi:
Tingkat kesalahan replikasi DNA jauh lebih rendah daripada transkripsi karena kebutuhan untuk melestarikan makna pesan genetik dari satu generasi ke generasi berikutnya. Misalnya, laju mutasi spontan pada E. coli adalah sekitar satu kesalahan per 10 10 basa yang digabungkan selama replikasi.
Hal ini terutama disebabkan oleh adanya bentuk tautomer minor dari basa yang telah mengubah sifat pasangan basa. Tingkat kesalahan diminimalkan dengan berbagai mekanisme. DNA polimerase hanya akan memasukkan nukleotida yang masuk jika membentuk pasangan basa yang benar dengan nukleotida cetakan di situs aktifnya.
Kesalahan sesekali dideteksi oleh exonuclease 3’—>5′ proofreading yang terkait dengan polimerase. Ini menghilangkan nukleotida yang salah dari ujung 3′ sebelum penggabungan lebih lanjut, memungkinkan polimerase kemudian memasukkan basa yang benar. Agar exonuclease proofreading berfungsi dengan baik, ia harus dapat membedakan pasangan basa yang benar dari yang salah.
Meningkatnya mobilitas pasangan basa ‘tidak terikat’ pada ujung 5′ paling akhir dari fragmen untai lagging DNA yang baru diinisiasi berarti bahwa mereka tidak akan pernah tampak benar sehingga tidak dapat dikoreksi. Oleh karena itu, beberapa nukleotida pertama adalah ribonukleotida (RNA) sehingga selanjutnya dapat diidentifikasi sebagai bahan fidelitas rendah dan diganti dengan DNA memanjang (dan mengoreksi) dari fragmen yang berdekatan. Kesalahan yang lolos dari proofreading dikoreksi oleh mekanisme perbaikan ketidaksesuaian.
Mekanisme Replikasi DNA pada Prokariota:
Replikasi DNA in vitro telah dipelajari secara ekstensif pada E.coli dan pada fag dan plasmid E.coli. Pada E. coli, proses replikasi DNA melibatkan tiga langkah utama berikut:
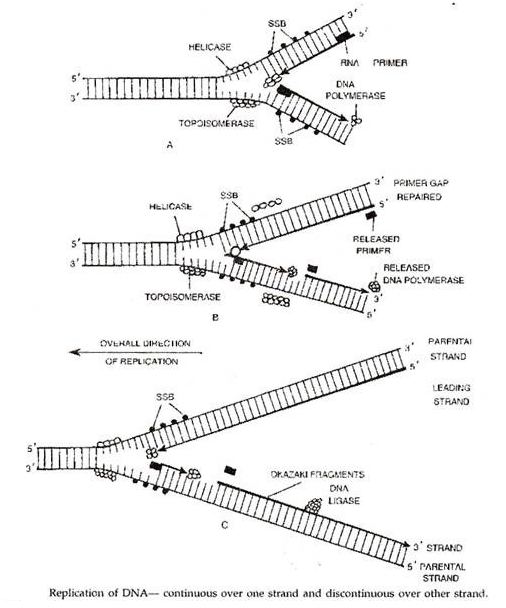
- Inisiasi replikasi DNA:
Proses ini terdiri dari tiga langkah: (i) pengenalan asal (O), (ii) pembukaan dupleks DNA untuk menghasilkan wilayah DNA beruntai tunggal, dan (iii) penangkapan protein Dna B (yaitu, 5′ 3′ helicase ; juga bertindak sebagai aktivator primase). Dengan demikian, kompleks ATP Dna-A (atau protein inisiator) berikatan pada 9 bp regio pengulangan terbalik (R,, R,, R v R 4 ) ori C dari E. coli dan mendorong pembukaan dupleks DNA pada regio tiga pengulangan langsung urutan 13-bp (disebut 13-mers).
Pembukaan terjadi dari kanan 13-mer ke kiri dan membutuhkan DNA superkoil negatif dan protein inisiator HU atau IHF. Dna B (-helicase) ditransfer ke DNA beruntai tunggal yang terpapar dan menyebabkan pelepasan DNA di hadapan A TP, protein SSB dan DNA gyrase (topoisomerase).
Ini menghasilkan pelepasan dupleks DNA dan replikasi dari ori C berlangsung di kedua arah (bidirectional); Pengikatan SSB terjadi pada daerah beruntai tunggal dan dua kompleks Dna B (=primosom) dimuat satu di setiap untai.
- Pemanjangan rantai DNA:
Langkah ini membutuhkan adanya enzim dan faktor berikut: 1. DNA B atau helicase (disebut juga mobile promotor); 2. primase (Dna G); 3. holoenzim DNA polimerase (atau DNA pol III HE); 4. protein SSB; 5. RNAase H yang menghilangkan primer RNA; 6. DNA polimerase I yang digunakan untuk mengisi celah yang tercipta karena primer RNA dan 7. DNA ligase (yang mengubah fragmen okazaki tanpa primer menjadi untai kontinu). Selama transisi inisiasi ke elongasi, peristiwa berikut terjadi:
(i) Saat helikase (atau Dna B) bergerak dalam arah 5′ —> 3′, ia menghasilkan garpu replikasi dengan membuka dupleks DNA.
(ii) Untai DNA yang memiliki helikase menjadi untai lagging. DNA primase berasosiasi dengan Dna B helicase, membentuk primosome yang mensintesis banyak primer untuk untai lagging dan primer RNA tunggal untuk untai terdepan.
(iii) Untuk sintesis untai lagging, DNA pol III HE harus bekerja pada untai yang sama dengan yang diikat oleh Dna B helicase, tetapi bergerak berlawanan arah.
(iii) DnaB helicase, Dna Gprimase dan DNA pol III FIE bekerja sama dalam pemanjangan untai. Rakitan helikase dan DNA polimerase tetap prosesif, yaitu tetap terikat erat pada garpu dan tetap terikat sepanjang reaksi.
Sintesis (- pemanjangan) untaian tertinggal dan terdepan terjadi dengan metode yang agak berbeda; itu jauh lebih kompleks untuk untai lagging daripada untuk untai terdepan:
(A) Sintesis terputus-putus pada untai lagging:
- Primase diambil dari larutan dan diaktifkan oleh helikase (DnaB) untuk mensintesis primer aRNA (panjang 10 hingga 20 nt atau nukleotida) pada untai lagging.
- Primer RNA dikenali oleh DNA pol III HE pada untai lagging dan digunakan untuk sintesis prekursor atau fragmen okazaki. Faktanya, setiap primer RNA baru dikenali oleh subunit gamma (y) dari DNA pol III HE dan diisi dengan subunit p dari polimerase yang sama. Subunit p yang dimuat sebelumnya ini kemudian dapat menangkap inti DNA poli III HE ketika tersedia setelah menyelesaikan pekerjaan sintetiknya pada fragmen okazaki sebelumnya.
- Setelah fragmen okazaki selesai, primer RNA dipotong oleh DNA polimerase 1, yang kemudian mengisi celah yang dihasilkan dengan DNA.
- Setelah DNA polimerase I menambahkan deoksiribonukleotida akhir pada celah yang ditinggalkan oleh primer yang dipotong, enzim ligase DNA membentuk ikatan fosfodiester yang menghubungkan ujung 3′ bebas pengganti primer ke ujung 5′ fragmen okazaki.
(B) Sintesis kontinyu pada untaian utama:
- Dalam replikasi DNA dua arah, untai terdepan diprioritaskan satu kali pada masing-masing untai induk.
- Primer RNA untai utama disintesis oleh enzim RNA polimerase.
- DNA pol III HE menyebabkan pemanjangan untai terdepan dan akhirnya enzim DNA pol 1 dan ligase memberikan sentuhan akhir pada untai terdepan seperti halnya untai tertinggal.
Replikasi DNA pada Eukariota :
Replikasi DNA eukariotik membutuhkan dua enzim DNA polimerase yang berbeda, yaitu DNA polimerase a dan DNA polimerase δ. DNA polimerase δ mensintesis DNA pada untai terdepan (sintesis DNA berkelanjutan), sedangkan DNA polimerase a mensintesis DNA pada untai tertinggal (sintesis DNA terputus-putus). Selain dua enzim tersebut, replikasi DNA: (1) antigen T; (2) faktor replikasi A atau RF-A (juga disebut RP-A atau SSB eukariotik); (3) topoisomerase I; (4) topoisomerase II; (5) proliferasi – antigen inti sel (PCNA, juga disebut siklin), dan (6) faktor replikasi Cor RF-C.
Proses replikasi DNA eukariotik melibatkan langkah-langkah berikut:
- Sebelum dimulainya sintesis DNA, terdapat tahap presintesis selama 8-10 menit untuk pembentukan kompleks DNA yang tidak terurai. Langkah ini hanya membutuhkan tiga protein murni, yaitu antigen T (T-ag atau antigen tumor), RF-A dan topiosomerase I dan II.
- T-antigen, menggunakan domain pengikat DNA-nya, membentuk kompleks multi-subunit dengan situs I dan situs II dengan adanya A TP dan menyebabkan pelepasan lokal.
- Pelepasan dupleks yang lebih luas terjadi karena asosiasi RF-A dan topoisomerase dengan – bantuan komponen DNA helicase dari T-ago Topoisomerase membantu pelepasan DNA dengan mengubah topologi DNA pada garpu replikasi.
- Protein RF-A atau SSB berikatan dengan DNA beruntai tunggal yang tidak terurai.
- Sintesis RNA primer dilakukan oleh primase yang berikatan erat dengan DNA polimerase ex.
- DNA polimerase a membantu sintesis fragmen okazaki dalam arah 5′ ke 3′.
- Faktor replikasi C (atau RF-C) dan PCNA (siklin) membantu pergantian polimerase DNA sehingga pol a digantikan oleh pol 5 yang kemudian terus mensintesis DNA pada untai terdepan.
- Fragmen okazaki lainnya kemudian disintesis dari garpu replikasi pada untai lagging oleh kompleks pol a – primase dan langkah ini diulangi lagi dan lagi, sampai seluruh molekul DNA tercakup.
- Primer RNA dihilangkan dan celah diisi seperti pada replikasi DNA prokariotik.
Baru-baru ini, peran DNA polimerase e dalam replikasi DNA telah ditekankan, sehingga tiga polimerase DNA (a, δ dan ε) sekarang diketahui terlibat dalam replikasi DNA eukariotik. A. Sugino dan rekan kerja telah mengusulkan bahwa DNA polimerase a mungkin berfungsi baik pada untaian terdepan dan tertinggal (karena polimerase a memiliki aktivitas α primase), sedangkan polimerase e dan polimerase 5 masing-masing terlibat dalam pemanjangan untai terdepan dan terbelakang.
Mekanisme Kerusakan dan Perbaikan DNA
Jenis Kerusakan DNA:
1. Lesi DNA:
Perubahan dalam struktur kimia atau fisik normal DNA disebut sebagai lesi DNA. Banyak agen eksogen, seperti bahan kimia dan radiasi, dapat menyebabkan perubahan posisi atom nitrogen dan karbon dalam sistem cincin heterosiklik dari basa dan beberapa gugus fungsi eksosiklik (yaitu gugus keto dan amino dari basa).
Hal ini dapat menyebabkan hilangnya pasangan basa atau pasangan basa yang berubah (misalnya A yang diubah dapat menjadi pasangan basa dengan C alih-alih T). Jika lesi seperti itu dibiarkan tetap berada di DNA, mutasi dapat terfiksasi dalam DNA melalui mutagenesis langsung atau tidak langsung.
Alternatifnya, perubahan kimia dapat menghasilkan distorsi fisik pada DNA yang menghalangi replikasi dan/atau transkripsi, menyebabkan kematian sel. Dengan demikian, lesi DNA mungkin bersifat mutagenik dan/atau mematikan. Beberapa lesi bersifat spontan dan terjadi karena reaktivitas kimia DNA yang melekat dan adanya spesies kimia reaktif yang normal di dalam sel.
Misalnya, basa sitosin mengalami deaminasi hidrolitik spontan untuk menghasilkan urasil. Jika dibiarkan tidak diperbaiki, urasil yang dihasilkan akan membentuk pasangan basa dengan adenin selama replikasi berikutnya, sehingga menimbulkan mutasi titik. Depurinasi adalah reaksi hidrolitik spontan lain yang melibatkan pemutusan ikatan N-glikosilat antara N-9 dari basa purin A dan G dan C-l’ dari gula deoksiribosa dan dengan demikian hilangnya basa purin dari DNA. Tulang punggung gula-fosfat DNA tetap utuh. Situs apurinik yang dihasilkan adalah lesi nonkode, karena informasi yang dikodekan dalam basa purin hilang.
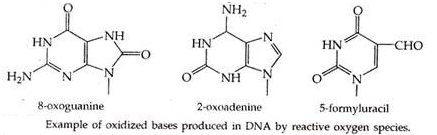
2. Kerusakan oksidatif:
Ini terjadi dalam kondisi normal karena adanya reactive oxy gen species (ROS) di semua sel aerobik, misalnya superoksida, hidrogen peroksida, dan yang paling penting, radikal hidroksil ( OH). Radikal ini dapat menyerang DNA, I pada sejumlah titik, menghasilkan serangkaian produk oksidasi dengan sifat II yang berubah, misalnya 8-oxoguanine, 2-oxoadenine dan 5-formyluracil. Tingkat ini dapat ditingkatkan oleh radikal hidroksil dari radiolisis air yang disebabkan oleh radiasi pengion.
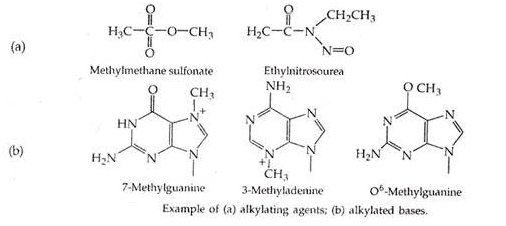
3. Alkilasi:
Zat alkilasi adalah bahan kimia elektrofilik yang dengan mudah menambahkan gugus alkil (misalnya metil) ke berbagai posisi pada asam nukleat yang berbeda dari yang dimetilasi oleh enzim metilasi normal. Contoh umum adalah metilmetana sulfonat (MMS) dan etilnitrosourea (ENU).
Contoh umum basa termetilasi adalah 7-metilguanin, 3-metil-adenin, 3-metilguanin dan 0 6 -metilguanin. Beberapa lesi ini berpotensi mematikan karena dapat mengganggu pelepasan DNA selama replikasi dan transkripsi. Sebagian besar juga secara tidak langsung bersifat mutagenik; namun, 0 6 -methylguanine adalah lesi mutagenik langsung karena dapat berpasangan dengan timin selama replikasi.
4. Adduct besar:
Dimer pirimidin siklobutana dibentuk oleh sinar ultraviolet dari pirimidin yang berdekatan pada satu untai dengan siklisasi atom karbon C5 dan C6 berikatan ganda dari setiap basa untuk menghasilkan cincin siklobutana. Hilangnya pasangan basa yang dihasilkan dengan untai yang berlawanan menyebabkan denaturasi lokal DNA menghasilkan lesi besar yang akan mengganggu replikasi dan transkripsi. Tipe lain dari dimer pirimidin, 6, 4-fotoproduk, dihasilkan dari pembentukan ikatan antara C6 dari satu basa pirimidin dan C4 dari basa yang berdekatan.
Ketika tar batubara karsinogen benzo[a]pyrene dimetabolisme oleh sitokrom P-450 di hati, salah satu metabolitnya (diol epoksida) dapat secara kovalen menempel pada gugus 2-amino dari residu guanin. Banyak agen arilasi aromatik lainnya membentuk adisi kovalen dengan DNA. Karsinogen hati aflatoksin B, juga secara kovalen berikatan dengan DNA.
Perbaikan DNA:
Sistem perbaikan mengenali berbagai perubahan dalam DNA untuk memulai tindakan. Sebuah sel mungkin memiliki beberapa sistem untuk menangani kerusakan DNA. Sistem ini meliputi: (1) Perbaikan langsung, (2) Perbaikan eksisi, (3) Perbaikan ketidaksesuaian, (4) Sistem toleransi, dan (5) Sistem pengambilan.
1. Perbaikan langsung:
Pembalikan atau penghilangan sederhana dari kerusakan DNA dikenal sebagai perbaikan langsung, misalnya penghilangan ikatan kovalen antara dua karbon 4 dan dua karbon 5 dari dua residu timin yang berpartisipasi dalam pembentukan dimer timin.
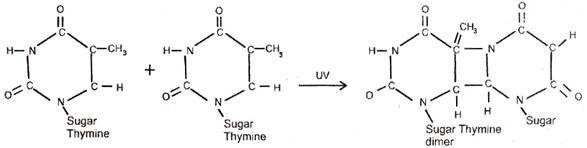
Dimer timin umumnya terbentuk karena radiasi UV dan mengganggu replikasi dan transkripsi. Kelner (1949) mengamati bahwa sejumlah besar bakteri dapat bertahan hidup dari radiasi UV dosis besar; jika mereka terkena sumber cahaya tampak yang tegang.
Fenomena ini disebut fotoreaktivasi. Belakangan diketahui bahwa selama penyinaran UV, suatu enzim tertentu terikat secara selektif pada DNA bakteri. Selama proses fotoreaktivasi, enzim diaktifkan oleh cahaya tampak. Itu membelah dimer timin, sehingga mengembalikannya ke bentuk aslinya. Proses perbaikan ini dimediasi oleh enzim dan bergantung pada cahaya.
2. Perbaikan eksisi:
Dalam mekanisme perbaikan ini, segmen untai DNA yang rusak atau berubah dipotong dan potongan DNA baru berukuran sintetik sebagai gantinya. Meskipun sistem perbaikan eksisi bervariasi dalam kekhususannya, jalur utama melibatkan tiga langkah berikut:
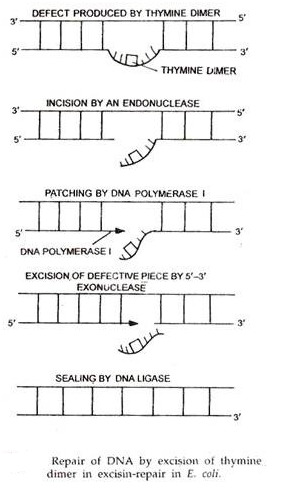
(i) Pengakuan dan Sayatan:
Bagian untai DNA yang rusak/diubah dikenali oleh endonuklease; enzim ini kemudian memotong untai yang terkena pada kedua sisi kerusakan.
(ii) Eksisi:
A 5′ –> 3′ exonuclease (DNA polimerase I) mencerna bagian yang rusak/berubah; ini menghasilkan wilayah beruntai tunggal dalam heliks ganda DNA.
(iii) Sintesis:
Pada langkah ini, daerah beruntai tunggal yang dihasilkan oleh eksisi berfungsi sebagai cetakan untuk DNA polimerase yang mensintesis pengganti segmen yang dipotong. DNA ligase kemudian menyegel nick yang tersisa setelah sintesis pengganti bagian yang dipotong.
Pada E. coli, eksisi kemungkinan besar disebabkan oleh aktivitas eksonuklease 3′–> 5′ dari DNA polimerase I, sementara sintesis dilakukan oleh aktivitas polimerase 5′–> 3′ dari enzim yang sama. Sistem perbaikan eksisi cukup umum baik pada prokariota dan eukariota. Beberapa dari sistem ini mengenali kerusakan umum pada DNA, sementara yang lain sangat spesifik, misalnya endonuklease AP menghilangkan residu ribosa dari tempat depurinasi. Perbaikan eksisi melibatkan panjang DNA yang berbeda dan dikelompokkan ke dalam tiga kelas berikut:
(a) Perbaikan tambalan sangat pendek (VSP):
Sistem ini berkaitan dengan perbaikan ketidaksesuaian antara pangkalan tertentu.
(b) Perbaikan tambalan singkat:
Dalam sistem ini sekitar 20 basa panjang untai DNA dipotong, dan kerusakan diperbaiki melalui gen uvr (uvr, A, B, C, dari E.coli), yang mengkode komponen endonuklease perbaikan. Enzim lain uvr D juga diperlukan untuk aktivitas helikase.
(c) Perbaikan tambalan panjang:
Sistem ini melibatkan pemotongan sekitar 1500 basis segmen panjang, tetapi terkadang segmen yang dipotong mungkin > 9000 basis. Ini jauh lebih jarang dan harus disebabkan oleh kerusakan yang menghalangi replikasi. Pada E. coli sistem ini juga melibatkan gen uvr dan DNA polimerase I.
3. Perbaikan Ketidakcocokan:
Ini melibatkan koreksi ketidaksesuaian atau pemasangan antara basis yang tidak saling melengkapi. Ketidakcocokan dapat timbul baik (a) selama replikasi atau (b) karena perubahan basa (misalnya, deaminasi sitosin menjadi urasil) dan menghasilkan distorsi struktural pada heliks ganda DNA. Pergantian ini ditangani di E. coli dengan sistem perbaikan eksisi tambalan sangat pendek yang dijelaskan di bawah ini.
Sistem Perbaikan Ketidakcocokan pada E. coli:
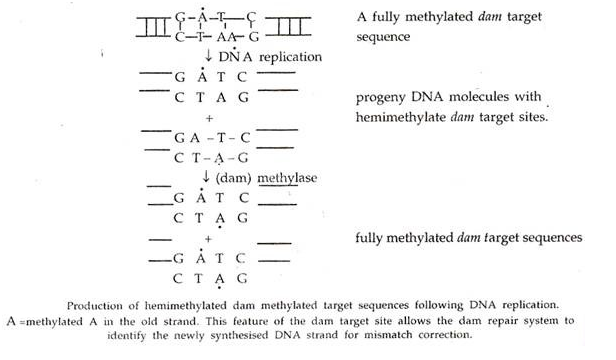
Ketika ada ketidakcocokan pada pasangan basa seperti pada GC -> GT, maka secara teoritis dapat diperbaiki untuk menimbulkan tipe liar (GC) atau tipe mutan (AT). Oleh karena itu, sistem perbaikan harus membedakan antara untaian lama dan baru dan hanya memperbaiki untaian baru untuk mengembalikan tipe liar.
Hal ini dilakukan dengan sistem perbaikan eksisi sangat pendek dan membutuhkan empat protein yaitu Mut L, Mut S, Mut U dan Mut H yang masing-masing dikodekan dalam E. coli oleh gen mut L, mut S, mut U dan mut H. dihasilkan selama replikasi diperbaiki oleh sistem perbaikan bendungan.
Bendungan gen E. coli menghasilkan metilase yang memetilasi adenin urutan GATC pada kedua untai DNA. Replikasi urutan GATC yang sepenuhnya termetilasi menghasilkan urutan hemimetilasi di mana residu A dalam untaian yang baru disintesis tidak termetilasi.
Keadaan non-metilasi dari urutan target ini digunakan untuk mengidentifikasi untai baru; basis di sekitar lokasi ketidaksesuaian pada untai baru dipotong dan penggantinya disintesis. Sistem ini melibatkan produk gen mut L, mut S, mut H, dan uvr D.
Perbaikan Sistem Pengambilan:
Sistem ini juga dikenal sebagai ‘perbaikan pasca-replikasi’ atau ‘perbaikan rekombinasi’. Pada E.coli, sistem perbaikan ini didasarkan pada gen rec A yang menghasilkan protein Rec A. Protein Rec A berfungsi dalam pertukaran untai antara molekul DNA selama rekombinasi genetik dan juga berfungsi dalam pertukaran untai tunggal selama perbaikan rekombinasi. Tampaknya ada dua jalur rec A, satu melibatkan gen rec B, C dan yang lainnya melibatkan gen rec F.
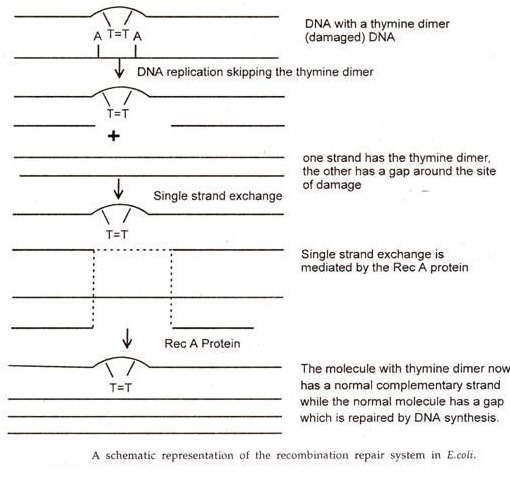
Sistem perbaikan ini beroperasi ketika distorsi struktural menghalangi replikasi di lokasi yang rusak. Misalnya, mutan E. coli yang kekurangan perbaikan eksisi tidak akan mampu menghilangkan dimer timin. Dalam situasi seperti itu, replikasi berlangsung secara normal hingga ke lokasi yang rusak; DNA polimerase menghentikan replikasi untai yang terpengaruh dan memulai kembali replikasi melewati situs yang rusak.
Untai pelengkap direplikasi secara normal di lokasi yang rusak di untai lainnya. Oleh karena itu replikasi menghasilkan satu progeni normal molekul DNA dan satu molekul yang memiliki dimer timin dalam satu untai dan celah panjang dalam untai komplementernya. Molekul DNA progeni dengan dimer timin akan hilang kecuali jika diperbaiki dengan mengisi celah di salah satu untaiannya.
Ini dicapai melalui pertukaran untai tunggal antara dua molekul DNA progeni; wilayah pertukaran mengisi celah dan diinduksi oleh protein Rec A. Sebagai hasil dari pertukaran ini, molekul progeni normal memiliki daerah beruntai tunggal yang memiliki celah. Kesenjangan ini diperbaiki dengan sintesis DNA.
Sistem Toleransi:
Sistem ini menangani kerusakan yang menghalangi replikasi normal di situs yang rusak mungkin dengan mengizinkan replikasi situs yang rusak dengan frekuensi kesalahan yang tinggi. Sistem ini mungkin sangat penting pada eukariota di mana ukuran genomnya sangat besar dan karenanya perbaikan kerusakan yang lengkap agak tidak mungkin.